Organismos Genéticamente Editados (1): Cultivos vegetales
En los años que llevo metido en el tema de bioseguridad, he notado que una de las principales razones para la oposición a los transgénicos por parte de los consumidores es la percepción que tienen sobre ellos.
Algunos consideran que transferir un gen de una especie (Ej: una bacteria o el ser humano) a otra (Ej: una planta o una vaca) no es un proceso natural. Pero es extraño que este prejuicio se desvanezca —o sea menos evidente— cuando se pregunta “¿y si el gen proviene de otro individuo de la misma especie o de una especie relacionada?”. A estos últimos algunos científicos le dan el nombre de “cisgénicos“. Un ejemplo son las papas genéticamente modificadas resistentes a la rancha que actualmente se vienen desarrollando en el Reino Unido. Estas papas —de la variedad Desiree— recibieron genes de resistencia a un hongo patógeno de una papa silvestre (Solanum venturii) usando las mismas técnicas empleadas para el desarrollo de una planta transgénica. Entonces, el hecho de que el gen provenga una especie emparentada, ¿lo hace “más natural”? La percepción de los consumidores indica que sí.
Por otro lado, los que conocen un poco más sobre cómo se hace un transgénico basan su rechazo en lo aleatorio que es la introducción de los transgenes en el ADN de un organismo, el cual podría provocar cambios indeseados en el metabolismo y fisiología de la planta. Los cierto es que las plantas transgénicas que actualmente se comercializan en el mundo han sido producidas con una tecnología desarrollada a finales de la década de 1970, la cual es para nada precisa. El transgén se puede incorporar en cualquier parte del genoma de la planta, incluso interfiriendo con genes importantes para su desarrollo. Sin embargo, estas “versiones fallidas” se desechan antes de llegar a comercializarse.
Aquí pueden ver una buena infografía (aunque no muy precisa) de cómo se hace un maíz transgénico hecha por Sergio Fernández.
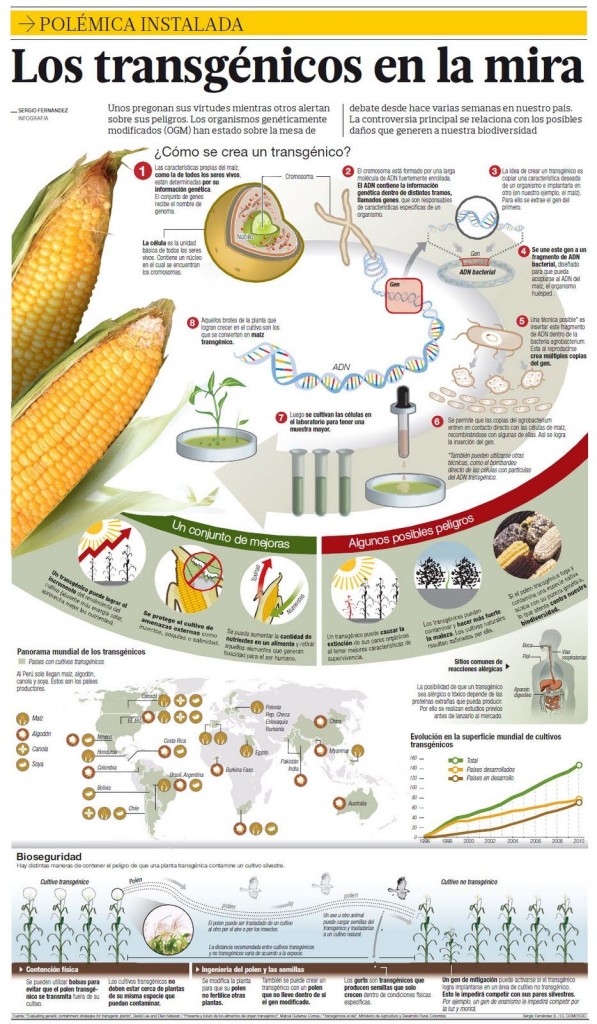
Dale click AQUÍ para ampliar.
Las plantas transgénicas que llegan a los campos de cultivo y a los mercados son aquellas que no presentan cambios aparentes respecto a sus contrapartes no transgénicas y, análisis genéticos a posteriori han permitido identificar en qué parte del genoma se ha introducido el (los) transgén(es). Desde un punto de vista reduccionista, está todo bien pues el transgén se ha incorporado en regiones no codificantes y muy estables del ADN de la planta, evitando así que interfiera con otros procesos celulares. Sin embargo, los avances en la genética de la última década han demostrado que esas regiones de ADN no codificante (mal llamado “ADN basura”), cumplen una importante función en la regulación de la expresión de otros genes.
En los últimos años, la ingeniería genética ha avanzado enormemente. Tal es así que hoy contamos con herramientas moleculares que podrían ayudarnos a superar los inconvenientes que presentan las plantas transgénicas: la transferencia de genes de una especie a otra y la aleatoriedad del proceso.
Edición genética
Los parientes silvestres de muchas plantas cultivadas presentan resistencia a plagas y enfermedades naturalmente. Incluso hay variedades que resisten mejor que otras al estrés ambiental como las sequías, el calor intenso o las heladas (cada vez más frecuentes debido al cambio climático). ¿Qué las hace diferentes si comparten casi los mismos genes entre sí? La respuesta es la variabilidad que pueda haber en esos genes. Unos cuantos cambios (mutaciones) en uno o más genes pueden darle a la planta una mejor respuesta a las condiciones adversas del entorno. Por lo tanto, una mayor diversidad genética (precisamente, lo que tenemos en el país) nos brindará más opciones para contrarrestar los efectos del cambio climático.
Hace unos años se publicó en la prestigiosa revista Nature la secuencia del genoma de la papa (trabajo en el que participó un grupo de investigadores peruanos). A partir de ese trabajo, el biólogo Roberto Lozano bajo la tutela de la Dra. Gisella Orjeda (actual presidenta del CONCYTEC) identificaron más de 400 potenciales genes de resistencia a enfermedades, de los cuales, casi la mitad estaban inactivos debido a pequeñas mutaciones.
Hoy en día, contamos con la tecnología para editar esos pequeños cambios y revertir las mutaciones, por lo que estaríamos en la capacidad de reactivar genes de resistencia en la papa y otras plantas cultivadas que ya cuentan con su genoma completamente secuenciado (Ej: arroz, tomate, maíz, trigo, etc.), sin necesidad de recurrir a genes procedentes de otros organismos.
Estas herramientas de edición de genes son las siguientes:
1. Las nucleasas dedos de zinc (ZNF, por sus siglas en inglés). Los dedos de zinc son unas estructuras proteicas que reconocen y se unen específicamente a una pequeña secuencia de ADN (tres pares de base por cada dedo). La nucleasa (Fok1), por su parte, es una proteína capaz de cortar las cadenas de ADN. Si juntamos estas dos proteínas tenemos una herramienta molecular capaz de hacer cortes en el genoma en lugares específicos.
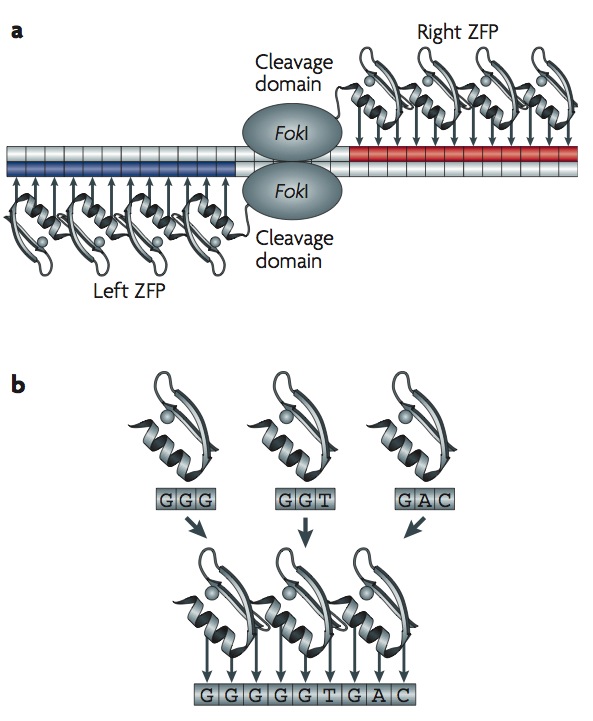
a) Los ZFN reconocen secuencias específicas de ADN para que la nucleasa actúe y realice cortes en el genoma de la planta. b) Cada dedo de zinc reconoce un triplete de nucleótidos. Fuente: Urnov et al. (2010) Nat Rev Genet.
2. TALEN. Esta herramienta molecular es muy parecida a las nucleasas dedos de zinc. La diferencia radica en que es mucho más específica porque cada estructura proteica cuenta con regiones repetidas de 34 aminoácidos, de los cuales el número 12 y 13 son los únicos que varían. Son precisamente estos dos aminoácidos los que le dan la especificidad por un determinado nucleótido del ADN, de acuerdo a un código identificado a fines del 2009.
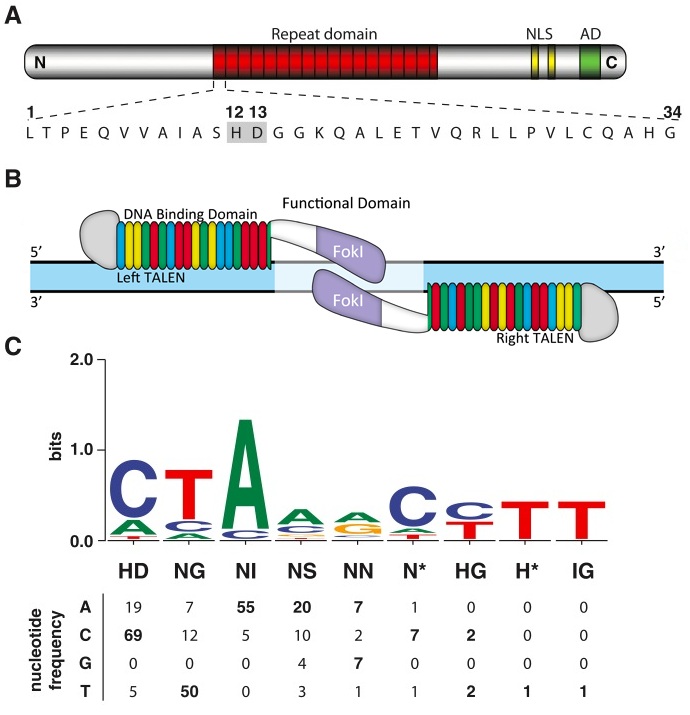
a) TALEN cuenta con un dominio de 34 aminoácidos que se repite varias veces (repeat domain) en los cuales los aminoácidos número 12 y 13 son los que varían. Cada letra representa un aminoácido específico b) El TALEN se une a una secuencia de ADN específica para que la nucleasa realice el corte en el genoma de la planta. c) En el 2009 se identificó qué combinaciones de aminoácidos 12 y 13 reconocen un nucleótido específico en el ADN. Fuente: Adaptado de Boch et al. (2009) Science.
Aquí un gráfico más sencillo sobre estas dos herramientas:

TALEN es más específico que ZFN porque reconoce las secuencias de ADN nucleótido por nucleótido. Fuente: Liu et al. (2013) Nat Rev Genet.
3. CRISPR/Cas9. Esta herramienta molecular utiliza una secuencia de ARN —en lugar de una estructura proteica— para reconocer la secuencia de ADN complementaria. Esta molécula de ARN se ubica dentro de la nucleasa Cas9 y la hace mucho más específica, eficiente y fácil de diseñar que los ZFN y TALEN.
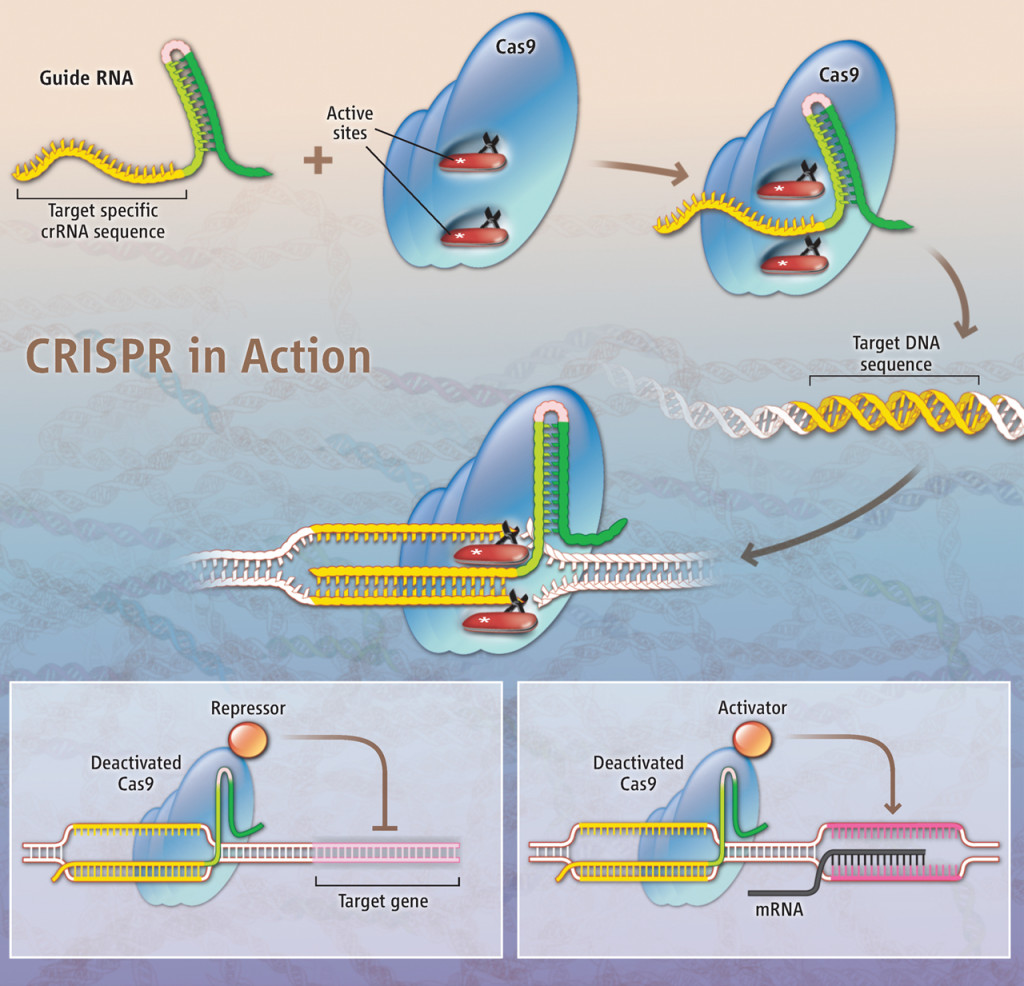
Sistema CRISPR/Cas9. La secuencia de ARN reconoce específicamente una secuencia complementaria de ADN para que actúe la nucleasa Cas9 y haga cortes en el genoma de la planta. Fuente: E. Pennisi. Science (2013). Credito: K. Sutliff/Science
Podemos aprovechar de todas estas herramientas para realizar cortes en el genoma y editar genes, ya sea quitando, añadiendo o cambiando fragmentos de ADN con el fin de revertir mutaciones y reactivar genes de importancia para la planta. Incluso podemos utilizarlas para introducir genes de otras especies en alguna región específica del genoma del organismo, evitando así la aleatoriedad de las herramientas empleadas en el desarrollo de las plantas transgénicas.
Estas nuevas herramientas de edición de genomas, especialmente el sistema CRISPR/Cas9, dará a los investigadores un mayor control a la hora de modificar y mejorar los rendimientos de los cultivos aprovechando las posibilidades que nos ofrece la diversidad genética con la que contamos en el país. Algunas pruebas en tabaco, sorgo y arroz han demostrado que la herramienta funciona.
Hay investigadores que aducen que las plantas desarrolladas por esta tecnología no deberían estar sujetas a las mismas regulaciones que las plantas transgénicas (incluso algunos dicen que ni siquiera deberían estar reguladas) pues no serían transgénicas ya que no se han introducido genes de otros organismos. Pero esto no sería del todo cierto ya que, en algunos casos, los genes que codifican los ZFN, TALEN o CRISPR/Cas9 deben introducirse dentro del genoma de la planta usando las herramientas empleadas en la producción de plantas transgénicas.
No obstante, las posibilidades que abre esta tecnología permitiría tener un mayor precisión al momento de hacer una modificación genética y se evitaría el uso de genes de otras especies para obtener mejores características.
¿Qué opinan ustedes? ¿Aceptarían consumir productos desarrollados por esta tecnología? ¿Lograría una mejor aceptación en los consumidores?
Notas:
TALEN significa “nucleasas efectoras tipo activador de transcripción”.
CRISPR significa “repeticiones palindrómicas cortas agrupadas y regularmente interespaciadas”.
Referencias:
Urnov, FD et al. Genome editing with engineered zinc finger nucleases. Nat Rev Genet 11: 636-646 (2010) doi: 10.1038/nrg2842
Liu, W et al. Advanced genetic tools for plant biotechnology. Nat Rev Genet 14: 781-793 (2013) doi: 10.1038/nrg3583
Nagamangala, C et al. Looking forward to genetically edited fruit crops. Trends Biotechnol. (2014) doi: 10.1016/j.tibtech.2014.07.003

:quality(75)/2.blogs.elcomercio.pe/service/img/expresiongenetica/autor.jpg)