¿Cómo se ve realmente una proteína?
Ayacucho, diciembre de 1996. Muchos niños hacíamos cola en la sede del rectorado de la Universidad Nacional San Cristóbal de Huamanga. Era la tarde en que se entregaban los regalos a los hijos de docentes y trabajadores de esta casa de estudios. Yo tenía 10 años y no pensé que ese día recibiría uno de los juguetes más memorables de mi vida. No era un Súper Nintendo ni un muñeco de colección de los Caballeros de Zodiaco (todos soñábamos con uno). Era… un microscopio.

Imagen referencial. El microscopio original todavía lo tengo en Ayacucho. Fuente: Amazon.
A pesar de ser un juguete venía con lentes de 100, 200 y 450 aumentos, lo mismo que los típicos microscopios del laboratorio de biología del cole. Con él podía observar claramente los pequeños organismos que viven en las aguas estancadas, células en gotas de sangre o en las alas de diversos insectos.
En la universidad ya utilizaba microscopios que amplificaban 1000 veces el tamaño de los objetos. Con estos podía observar los cromosomas de las células y todos los pasos de la división celular. Más adelante, utilicé microscopios de fluorescencia en los que se veían diversas estructuras celulares con diversos colores brillantes: azul, rojo o verde.
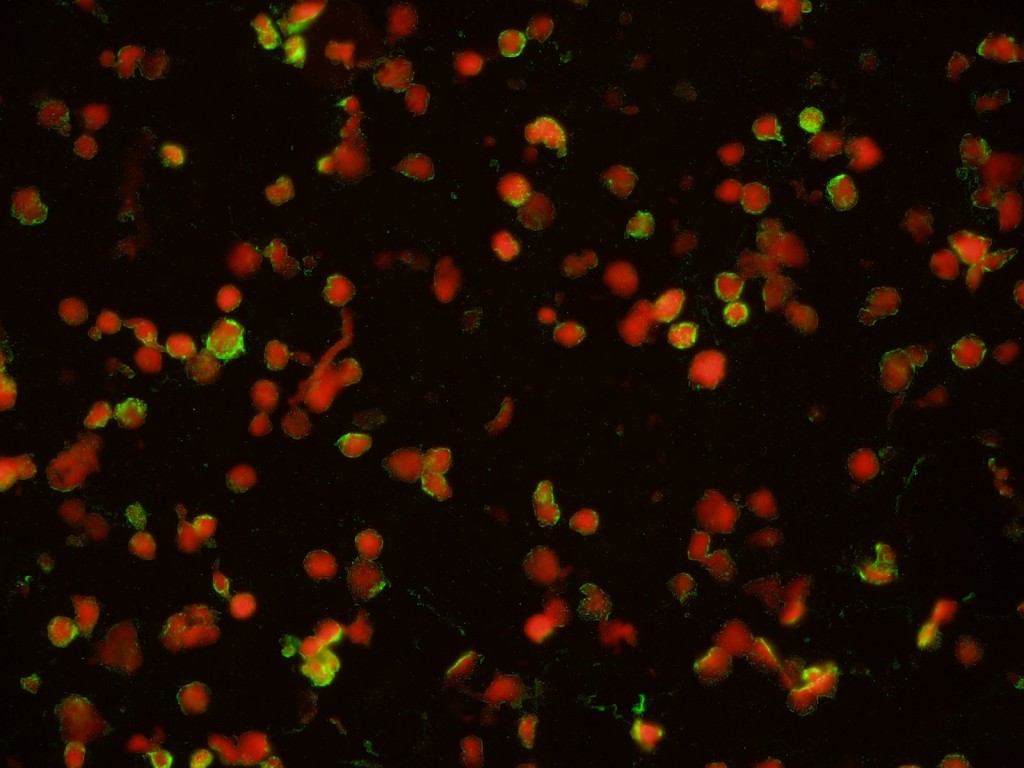
Celulas (rojas) infectadas por Mycoplasma pneumoniae (verde) a través de inmunoinfluorescencia indirecta. Fuente: Vircell.
Sin embargo, este tipo de microscopía tiene un límite. Cosas tan pequeñas como los virus, que tienen la millonésima parte del volumen de una célula, ya no podían ser observadas. Esto se debe a que a la naturaleza de la luz.
Si hacemos un poco de memoria, la luz es una onda cuya distancia entre cresta y cresta (longitud de onda) esta comprendida entre los 400 y 700 nanómetros (nm). Entonces, si queremos ver cosas que miden menos de 400 nm —como los virus que miden unos 50 nm— la luz no podrá distinguirlas. Se requiere de una “luz” con longitudes de onda más pequeñas.

Espectro electromagnético. Fuente: SCENIHR.
En la década de 1920 se supera este inconveniente utilizando rayos de electrones —con longitudes de onda muy chiquitas— en vez de luz. Así nace la microscopía electrónica y con ella hemos podido revelar la forma de los virus, los detalles de la superficie de las células, la estructura de las mitocondrias y cloroplastos, etc.
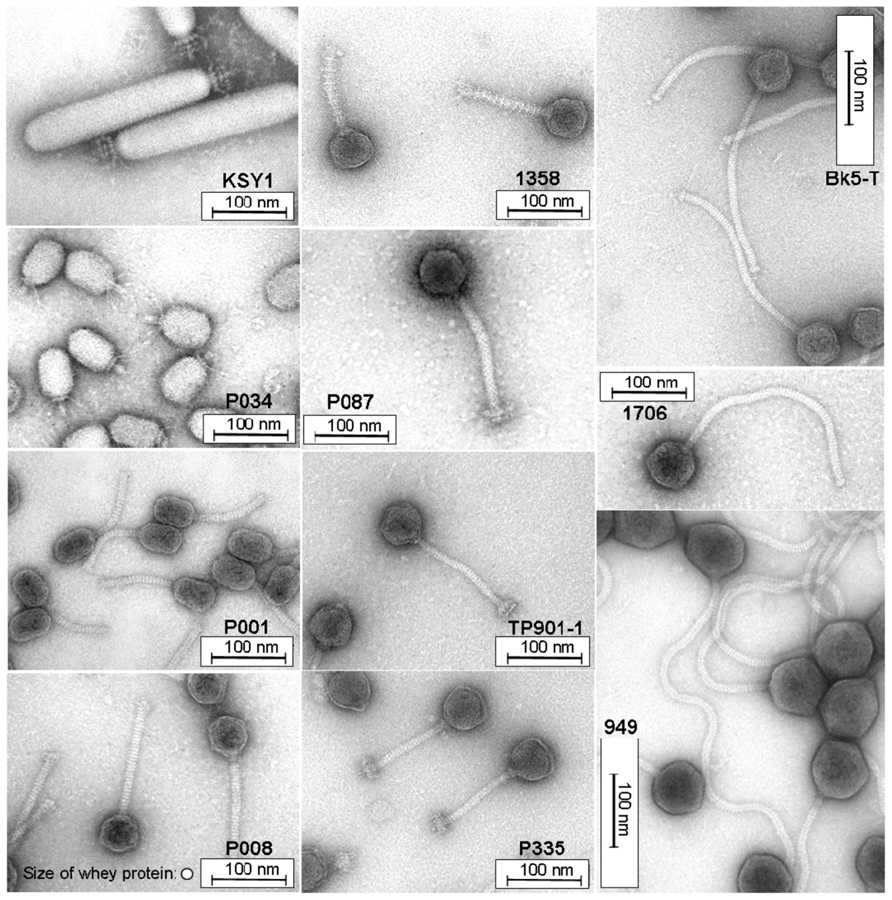
‘Selfies’ de diferentes tipos de virus. Fuente: Atamer, Z. et al (2013).
Sin embargo, los científicos no se conformaban con eso. Querían ver cosas aún más pequeñas como la estructura de las proteínas o del ADN con tamaños que rondan los 5 nm. Para que te hagas una idea de lo chiquito que son, imagina que todo el Estadio Nacional es una célula. A esta escala, las proteínas no serían más grandes que canicas o pelotas de golf. La microscopía electrónica ya no permitía llegar a ese nivel de detalle.
Entre la década de 1920 y 1960 empezó a desarrollarse una técnica para determinar la estructura de diversas moléculas complejas, entre ellas, las proteínas. El método se basaba en el uso de los rayos X, cuya longitud onda va de 0,1 a 10 nm.
Cuando los rayos X atraviesan una molécula se desvían de acuerdo a la posición de sus átomos. Esto genera un patrón de difracción que, posteriormente, es analizado con algoritmos muy complejos —que hoy en día lo hacen las computadoras— para revelar la forma y estructura de la molécula en estudio. Pero, para realizar esta técnica, la molécula debe estar cristalizada, es decir, deben estar organizadas formando una estructura sólida, uniforme y estable. Por ejemplo, el hielo es la forma cristalizada del agua. La sal y el azúcar de mesa son las formas cristalizadas del cloruro de sodio y la sacarosa, respectivamente.
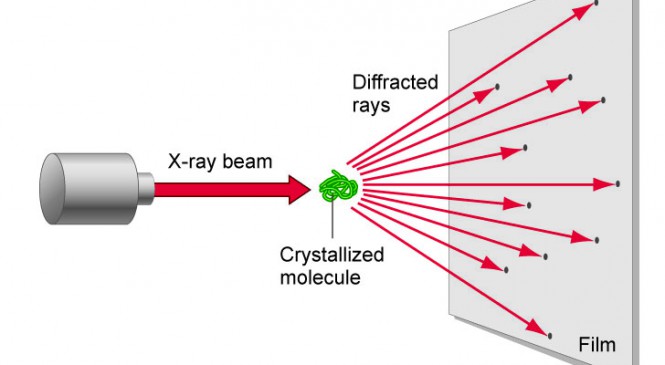
Difracción de rayos X. Fuente: The Rogues’ Gallery.
El problema es que muchas moléculas biológicas como las proteínas no pueden ser cristalizadas. A parte, los rayos X, al tener una longitud de onda más corta, son sumamente energéticos. Esto provoca la destrucción de la proteína mucho antes de poder revelar su estructura. Y, además, los cristales están compuestos por millones de proteínas por lo que unas interfieren con otras, provocando que las imágenes salgan borrosas. Por esta razón, es necesario desarrollar una metodología para poder observar directamente la forma de proteínas individuales.
Después de años de trabajo, un grupo de investigadores del Departamento de Física de la Universidad de Zurich, lo consiguieron según un artículo prepublicado el 30 de diciembre pasado en ArXiv.
Lo que hicieron fue rociar una solución de tres proteínas: el citocromo C (CytC), la albúmina de suero bovina (BSA) y la hemoglobina (Hb), sobre una lámina de grafeno en una cabina de ultravacío para evitar cualquier interferencia. Luego pusieron la lámina con la muestra en un microscopio electrónico de holografía de baja energía.
Lo que hace este microscopio es emitir un rayo de electrones de baja energía (para evitar dañar la muestra) sobre las proteínas. Luego, detectan la interacción (rebotes y desvíos) de estos electrones con la molécula en cuestión generando un holograma. Finalmente, una computadora usa esta imagen holográfica para reconstruir la estructura original de la proteína. Se pueden usar imanes para obtener otros ángulos. Aquí un video de cómo funciona esta técnica.
Aquí un esquema que resume el trabajo:

El rayo de electrones de baja energía incide sobre la proteína depositada en la lámina de grafeno que, por su grosor (un átomo), es transparente. Fuente: Longchamp et al. (2015) ArXiv.
Y, ahora prepárense que verán por primera vez como son realmente tres distintas proteínas:
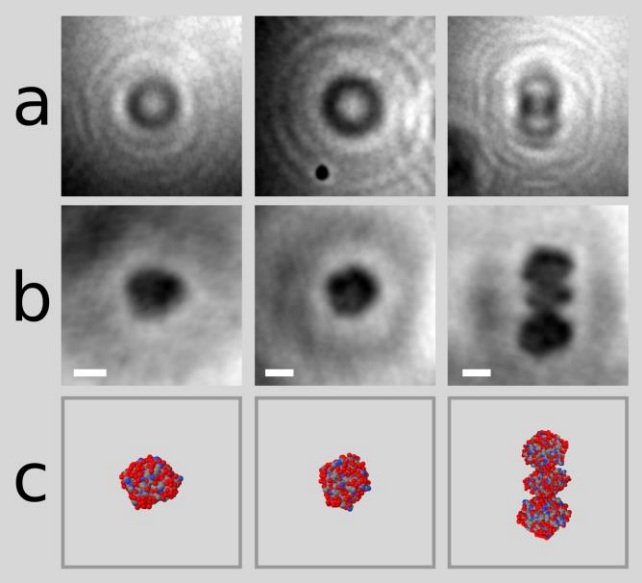
Foto real de un Citocromo C. a) los hologramas, b) la reconstrucción en computadora y c) la estructura determinada anteriormente por rayos X. Escala 2 nm. Fuente: Longchamp et al. (2015) ArXiv.

Foto real de la Hemoglobina C. a) los hologramas, b) la reconstrucción en computadora y c) la estructura determinada anteriormente por rayos X. Escala 5 nm. Fuente: Longchamp et al. (2015) ArXiv.
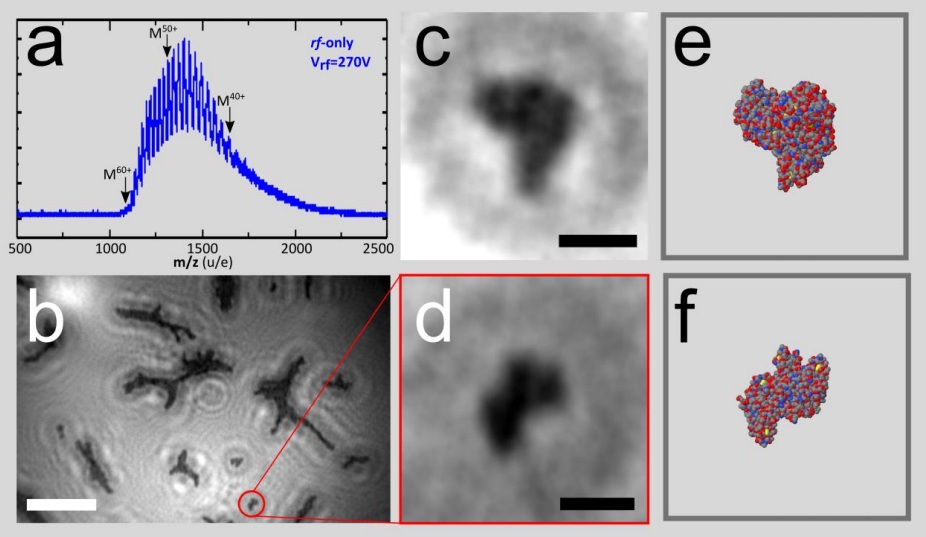
Albúmina de suero bovino, paso a paso. a) espectro de masas de BSA, b) BSA depositado sobre la lámina de grafeno, c) y d) imágenes de la BSA en distintas posiciones, e) y f) estructura determinada anteriormente por rayos X de las imágenes c) y d). Fuente: Longchamp et al. (2015) ArXiv.

:quality(75)/2.blogs.elcomercio.pe/service/img/expresiongenetica/autor.jpg)