Desarrollan bacteria con ADN de seis letras
A, T, G y C. Toda la vida en la Tierra está escrita (o codificada) usando sólo estas cuatro letras que representan a cada uno de los componentes básicos del ADN —los nucleótidos Adenina, Timina, Guanina y Citosina.
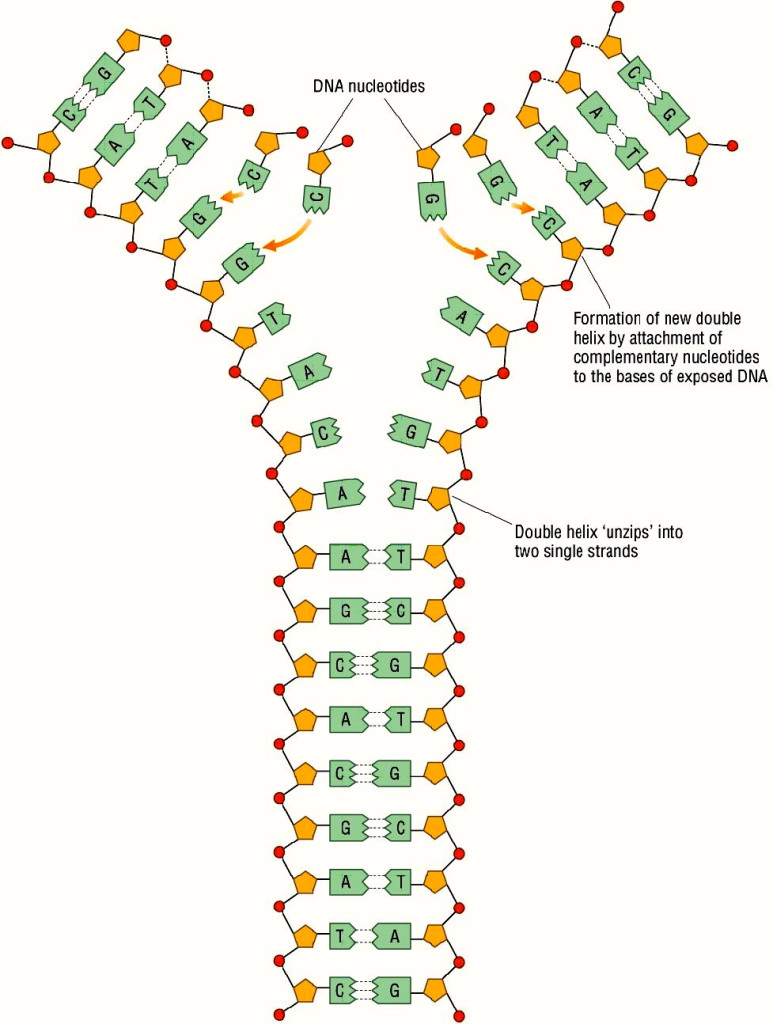
Replicación de ADN en función a la complementariedad de las bases. Fuente: Internet.
El ADN es una larga cadena de nucleótidos en forma de una escalera de caracol. Presenta dos hebras que se unen de manera complementaria: A:T y C:G. La longitud y la secuencia de la cadena (…TAGCATGACAAA…) es específica de cada especie, con ligeras variaciones entre un individuo y otro. Básicamente, el ADN almacena la información requerida para generar un determinado ser vivo. Y, gracias a la complementariedad entre los nucleótidos, la información puede ser transmitida de generación en generación de manera fidedigna, utilizando una de las hebras como molde.
Se dice que el ADN almacena y transmite información codificada porque la lectura de la secuencia se hace en tripletes (…TAG-CAT-GAC-AAA-…), donde cada combinación cifra para un aminoácido específico, de acuerdo a la siguiente tabla:
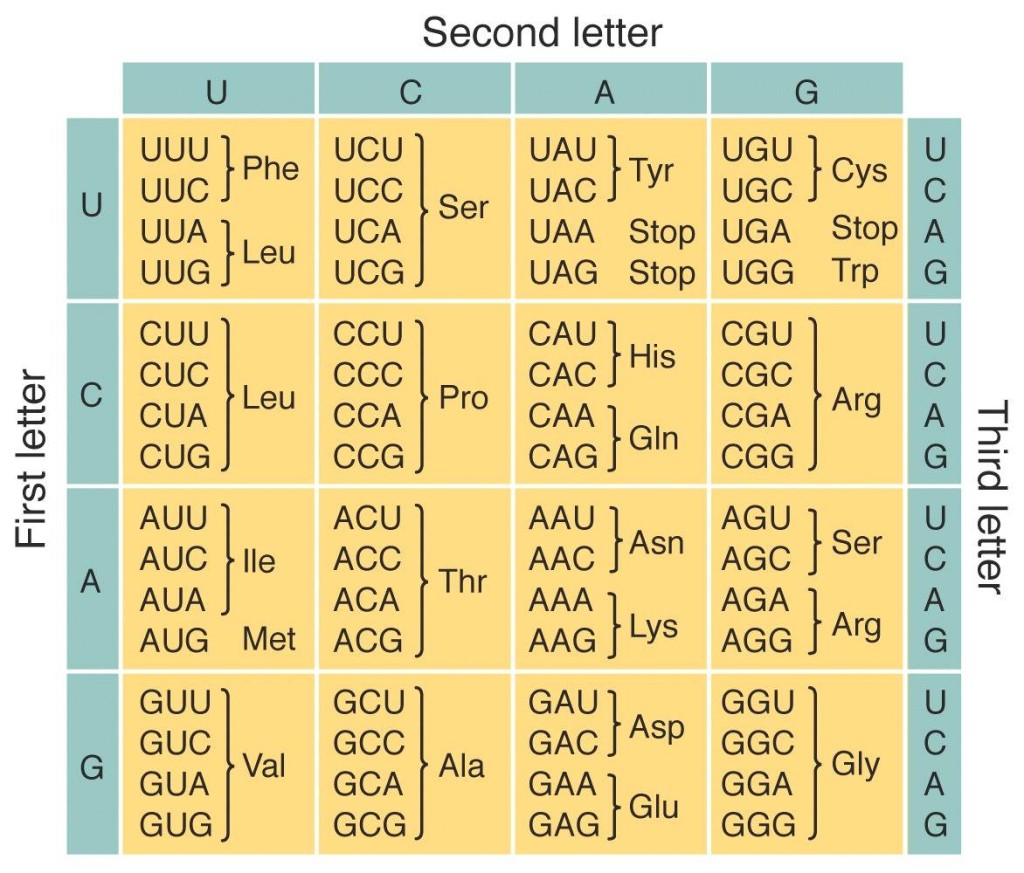
El código genético. Las Timinas (T) son reemplazadas por Uracilo (U) al momento de transcribir la información del ADN a ARN. Fuente: Internet.
Las 64 combinaciones posibles codifican los 20 aminoácidos escenciales para la vida. Con ellos se construyen todas las proteínas que permiten el desarrollo de un ser vivo, pues cumplen con importantes funciones: forman la estructura y soporte de las células, y facilitan la síntesis de todos los componentes esenciales para su funcionamiento.
¿Qué pasaría si introducimos dos letras más —X e Y— al alfabeto de la vida? Ampliaríamos el número de combinaciones de tripletes disponibles, pasando de 64 a 216. Esto —en teoría— abriría la posibilidad de incorporar nuevos aminoácidos al lenguaje de la vida para generar proteínas con funciones biológicas que no existen en la naturaleza. Incluso, se podrían generar nuevas formas de vida.
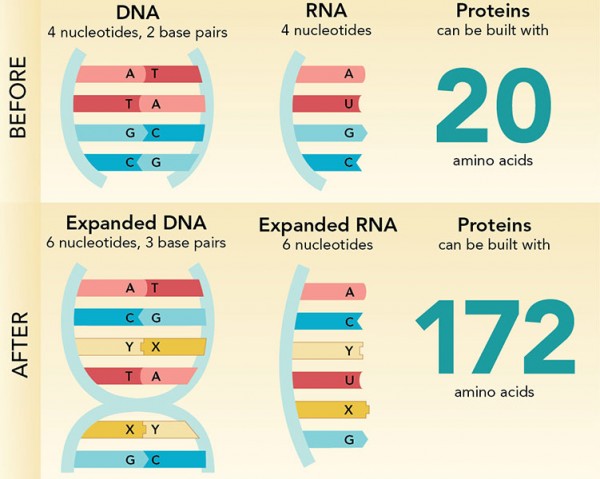
Las posibilidades que abriría la incorporación de un nuevo par de nucleótidos en el ADN. Fuente: Science.
Hace un par de años, un grupo de investigadores del The Scripps Research Institute, liderados por el Dr. Floyd Romesberg, lo lograron. Incorporaron los nucleótidos d5SICS y dNaM (X e Y) en el ADN de una bacteria, la cual pudo vivir y transferir dicho material genético a sus descendientes. No obstante, el microorganismo “enfermaba”. Tardaba más tiempo en dividirse y perdía los nucleótidos X e Y después de unas pocas generaciones.
Es así que Romesberg y su equipo investigaron qué era lo que pasaba. Su objetivo ahora era lograr la estabilidad de los nuevos nucleótidos en el ADN para que se mantengan de forma indefinida.
Lo primero que descubrieron fue que una porción de la proteína encargada de transportar los nucleótidos X e Y hacia el interior de la bacteria, era la responsable de su intoxicación. Al rediseñar la proteína sin dicha porción, la bacteria recuperaba su viabilidad original —ya no tardaba tanto en dividirse.
También observaron que los nucleótidos sintéticos X e Y se perdían porque las enzimas responsables de replicarlas (polimerasas) cometían errores: o bien los reemplazaban por nucleótidos naturales (ATCG) o bien los eliminaban. Para ello, cambiaron el d5SICS por dTPT3. Ahora la replicación era óptima porque los nuevos nucleótidos eran reconocidos de manera más eficiente por las polimerasas.
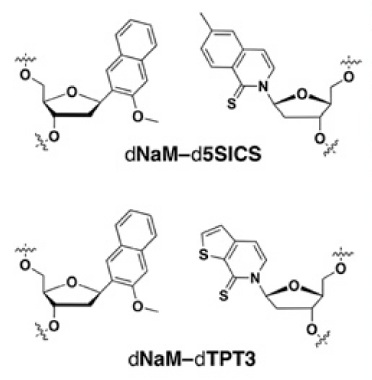
Arriba: Los primeros nucleótidos X e Y utilizados en el 2014. Abajo: los nucleótidos X e Y optimizados en el presente trabajo. Fuente: Zhang et al. (2017).
Finalmente, diseñaron un sistema CRISPR-Cas9 que reconocía y degradaba los fragmentos de ADN que perdían o cambiaban los nucleótidos sintéticos X e Y, para quedarse solo con aquellos que los poseían.
Con todos estos pequeños ajustes, Romesberg y su equipo lograron retener los nucléotidos sintéticos en el 100% de los descendientes hasta por 60 generaciones. Además las bacterias mejoraron su viabilidad y su tasa de multiplicación solo se redujo en un 17%.
Los investigadores dejan en claro que todavía no hay ninguna aplicación para este tipo de bacterias con ADN semisintético. El siguiente paso es lograr que un triplete con el nucleótido X o Y codifique uno de los 20 aminoácidos esenciales. De esta manera, sentar las bases para expandir el código genético y generar proteínas que incorporen otros aminoácidos sintéticos, con funciones biológicas novedosas y diversas aplicaciones.
Referencia:
![]() Zhang, Y., Lamb, B., Feldman, A., Zhou, A., Lavergne, T., Li, L., & Romesberg, F. (2017). A semisynthetic organism engineered for the stable expansion of the genetic alphabet Proceedings of the National Academy of Sciences DOI: 10.1073/pnas.1616443114
Zhang, Y., Lamb, B., Feldman, A., Zhou, A., Lavergne, T., Li, L., & Romesberg, F. (2017). A semisynthetic organism engineered for the stable expansion of the genetic alphabet Proceedings of the National Academy of Sciences DOI: 10.1073/pnas.1616443114

:quality(75)/2.blogs.elcomercio.pe/service/img/expresiongenetica/autor.jpg)