Bacteria salvada por proteína sintética
A, T, C y G. ¿Qué podrías hacer con estas cuatro letras? ¿Acaso escribir un poema o una canción? No creo. Como sea que las combines no podrás formar más de una decena de palabras. A lo mucho te alcanzará para un reguetón: “Gata, ataca, taca taca ta (repetir 16 veces)”. Sin embargo, lo que ha hecho la evolución con ellas es asombroso. Ha utilizado estos cuatro caracteres para almacenar y transmitir información, de generación en generación y por casi 4000 millones de años, de forma codificada, a través de una molécula maravillosa llamada ADN. Las cuatro letras representan sus cuatro componentes básicos: Adenina, Timina, Citosina y Guanina.
Sin embargo, te preguntarás ¿qué tipo de información se puede almacenar con ella? Pues nada menos que las instrucciones necesarias para “construir” un ser vivo cualquiera, desde una diminuta hormiga hasta un voluminoso elefante.
ATCG vendría a ser como el código binario (1, 0) de las computadoras. No sirven de mucho si no se traduce a un lenguaje funcional. En el caso de los seres vivos, este lenguaje está hecho en base a veinte aminoácidos. Entonces ¿cómo hacemos para que a partir de cuatro caracteres iniciales codifiquemos un lenguaje funcional basado en veinte? Fácil, agrupándolos y combinándolos.
Si los agrupamos de dos en dos (AA, AG, TA, GG, CT, etc.), sólo obtendremos 16 combinaciones. No alcanza. Pero si los agrupamos en tripletes (ATC, CTT, GAC, TTT, etc.), obtendremos 64 combinaciones. Eso es más que suficiente, incluso sobran. Para evitar esto, algunos aminoácidos son codificados por más de una de estas combinaciones. A esto le llamamos el código genético y lo conocemos desde hace más de 50 años.
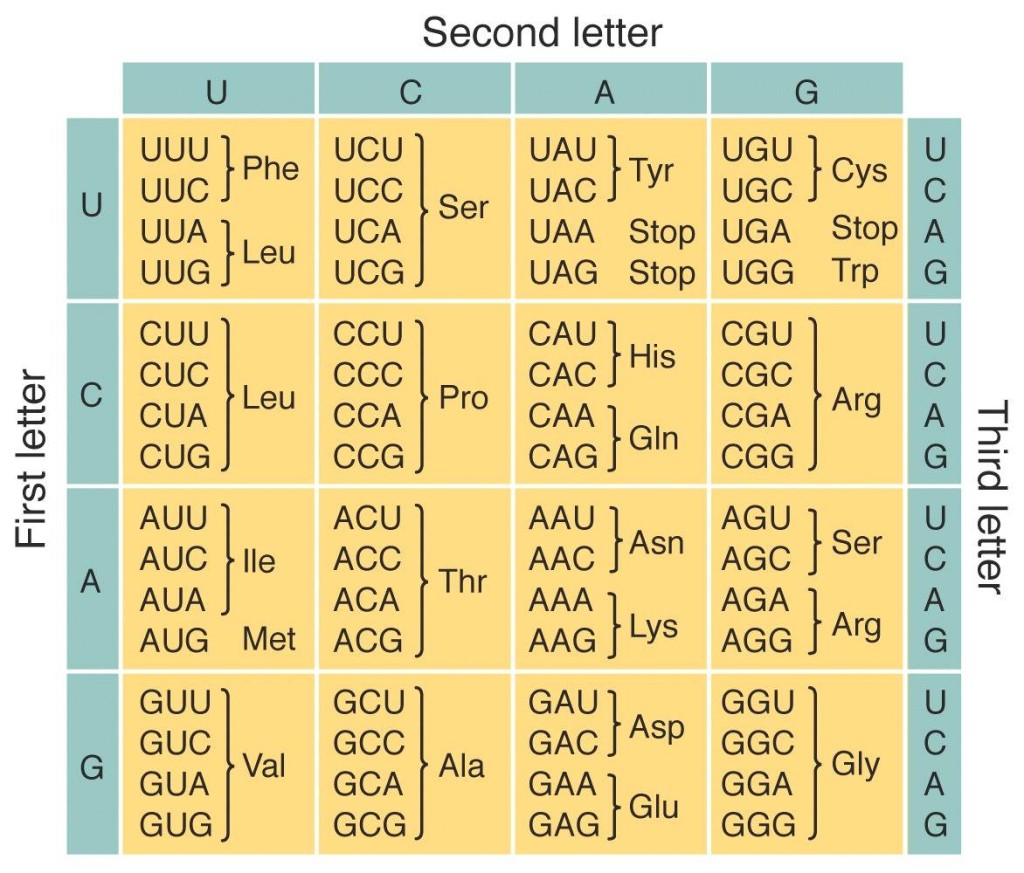
El código genético. Los 20 aminoácidos y tres secuencias “STOP” que indican dónde finaliza la traducción de la secuencia. Fuente: Geek.
Y ¿de dónde salió la U? La respuesta es que la secuencia de ADN no se traduce directamente en una secuencia de aminoácidos. Antes es transcrita en una secuencia de ARN que lleva la información codificada desde núcleo de la célula (o nucleoide en caso de las bacterias), hasta los ribosomas, que son la maquinaria molecular responsable de realizar la traducción. Cuando la información pasa del ADN al ARN, las timinas (T) son reemplazadas por uracilo (U).
A partir de una secuencia de ADN como …ATCGTAGCTACG… podemos generar una secuencia de aminoácidos …Ile-Val-Ala-Thr… que pueden ser péptidos (si son cortas) o proteínas (si son largas). Estas secuencias no son aleatorias, sino que han sido moldeadas por millones de años de evolución para realizar funciones específicas en los seres vivos. Entonces, en términos generales, a toda secuencia de ADN que codifica una secuencia de aminoácidos funcional le llamamos gen.
El número de combinaciones posibles con 20 aminoácidos es inmensa. Por ejemplo, habría 5 x 10132 (eso es un 5 seguido por 132 ceros) posibilidades de construir una pequeña proteína de 102 aminoácidos. Eso es unas 1052 veces el número de átomos en el universo.
Un diminuto microbio como E. coli tiene unos 4300 genes. En el laboratorio puede vivir muy bien en un medio de cultivo con un poco de glucosa (azúcar) y algunos micronutrientes (p. ej.: fósforo, nitrógeno, magenesio, entre otros). Sin embargo, si se le quita el gen que codifica la proteína serB, muere a menos que le incorporemos serina —un aminoácido esencial, que forma parte de otras proteínas— al medio de cultivo. Es decir, serB es esencial para E. coli.
En el 2011, Michael Hecht, investigador de la Universidad de Princeton (EEUU), utilizó el código genético para crear más de un millón de secuencias aleatorias de aminoácidos. Luego, introdujeron los genes que codificaban estas proteínas sintéticas en distintas cepas de E. coli para ver si alguna de ellas podría cumplir algún tipo de función dentro del microorganismo.
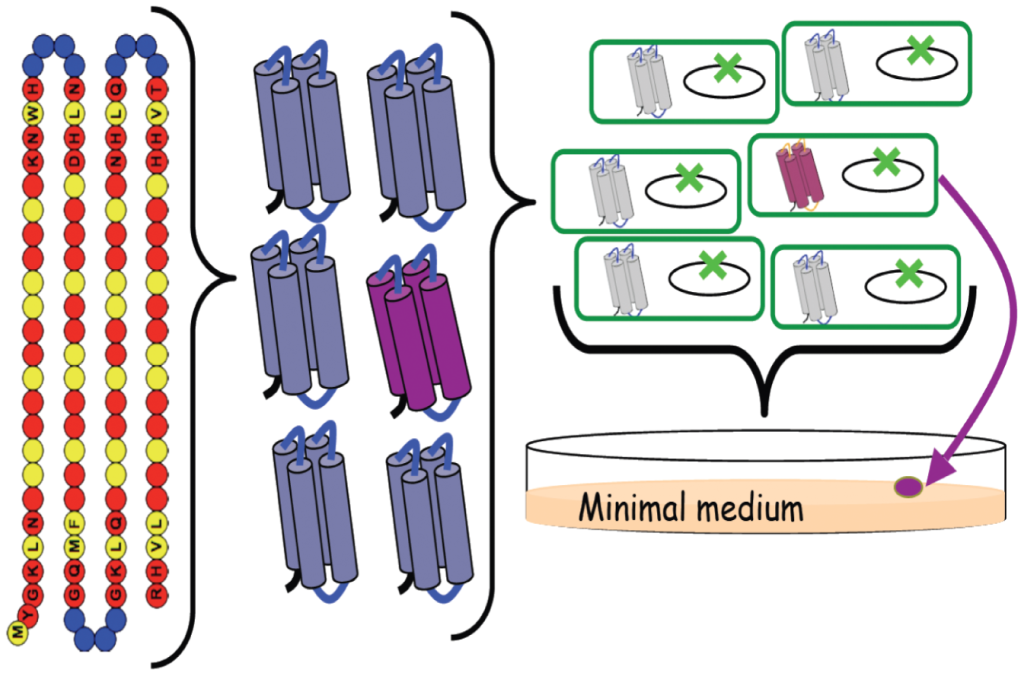
Proteínas sintéticas desarrolladas por Hecht y colaboradores. Aminoácidos polares (rojo) y no polares (amarillo). Fuente: Hecht et al (2011) PLOS One.
Hecht y su equipo identificaron una proteína sintética que le permitía sobrevivir a la cepa de E. coli carente del gen serB en un medio sin serina, por ello la llamaron SynSerB3. Pero ¿cómo lo hacía?
Contra todo pronóstico SynSerB3 no reemplazaba la función de la SerB original. A pesar de ser una proteína desarrollada artificialmente y que no ha pasado por un proceso evolutivo, su función era más compleja: regulaba la expresión de más de 600 genes, especialmente, el gen hisB que se volvió 10 veces más activo de lo normal.
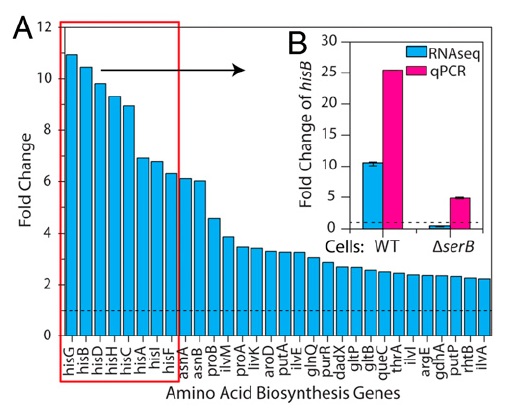
Aumento en los niveles de expresión de distintos genes involucrados en la síntesis de aminoácidos. Fuente: Digianantonio & Hecht (2016) PNAS.
HisB codifica una proteína (enzima) involucrada en la síntesis de la histidina (otro aminoácido esencial para la bacteria). Sin embargo, los investigadores descubrieron que HisB también podía sintetizar la serina (tenía una función “promiscua”), rescatando así a la E. coli sin SerB de la muerte.
Este trabajo es un gran avance en el campo de la biología sintética. Una proteína diseñada de manera aleatoria en el laboratorio y que no existen en la naturaleza, ha demostrado ser capaz de cumplir funciones vitales en un ser vivo. Dada las infinitas posibilidades para combinar los aminoácidos, podemos generar un arsenal de proteínas sintéticas con funciones nunca antes vistas en la naturaleza. Las aplicaciones de la biología sintética se pueden dar en todos los campos de la actividad humana. Sin embargo, nunca se debe dejar de lado la bioseguridad al realizar estos experimentos.
Referencia:
Digianantonio & Hecht. PNAS February 16, 2016, doi: 10.1073/pnas.1600566113 (2016)

:quality(75)/2.blogs.elcomercio.pe/service/img/expresiongenetica/autor.jpg)