Ahora vienen los Organismos Genéticamente Recodificados (OGR)
La semana pasada, la revista Nature publica dos estudios revolucionarios que han sido abordados por los principales medios de comunicación científica: Discover, Nature News, The New York Times, The Loom (una de las mejores historias), NPR, The Guardian, entre otros. En español, lo más recomendable está en El Mundo y El País de España.
¿De qué se trata? Pues, dos grupos de investigadores —uno de Yale y otro de Harvard— han creado bacterias genéticamente modificadas que requieren de un aminoácido sintético (que no se puede encontrar en la naturaleza) para poder vivir.
Crédito: Spencer Katz.
Diseminación de transgénicos
Desde que se crearon los primeros Organismos Genéticamente Modificados (OGM o transgénicos), allá por la década de 1970, hubo cierta preocupación por parte de la comunidad científica de que estos se diseminen fuera de control, invadiendo nuevos ecosistemas y desplazando o afectando a las especies nativas.
Diversas estrategias de contención se han desarrollado. Por ejemplo, una de ellas incorpora en el organismo genes letales que se activan si no está presente el antídoto —un nutriente específico— en el medio. Otra estrategia convierte al organismo dependiente de una molécula específica para vivir, algo así como en la película Crank donde el protagonista debe mantener los niveles de adrenalina altos en su cuerpo para no morir a causa del veneno. Pero la estrategia más usada es quitar un gen que produce alguna molécula esencial para vivir, por lo que esta debe ser incorporada siempre en la dieta del organismo sino muere. A estos últimos se les llama auxótrofos.
Todos los seres vivos somos auxótrofos a alguna molécula esencial. Por ejemplo, los humanos no podemos producir la vitamina C o la vitamina A, así que requerimos de ellas en nuestra dieta. De no obtenerlas, sufrimos de graves enfermedades a causa de su deficiencia (escorbuto y ceguera, respectivamente).
Sin embargo, la auxotrofía no es segura. El organismo puede adquirir la molécula esencial de otra fuente o mutar alguno de sus genes para volver a producirla.
En el 2009, un científico llamado Malcolm Casadaban fue infectado en el laboratorio por una cepa de Yersinia pestis (bacteria que produce la temible peste). Esta bacteria fue modificada genéticamente para que dependiera de hierro libre en el medio para poder vivir. Normalmente, Y. pestis es capaz de ‘robar’ este elemento de la hemoglobina de nuestra sangre; así que los científicos le quitaron los genes que le daban esta habilidad para poder trabajar con ella sin ningún riesgo. Sin embargo, Malcolm tenía una extraña enfermedad no diagnosticada llamada hemocromatosis, cuya característica es —para mala suerte— tener grandes cantidades de hierro libre en el cuerpo. El científico murió. ¿Quién lo diría? Un hecho considerado como muy improbable ocurrió. La medida de seguridad no funcionó.
Otro ejemplo son los famosos peces fluorescentes. Estos animalitos poseen genes de anémonas y medusas que codifican proteínas que brillan si son puestos bajo luz azul o UV. Son bonitos para tenerlos en una pecera. Para evitar que se reproduzcan y diseminen, los científicos los desarrollan estériles o de un mismo sexo (puros machos). Sin embargo, un investigador peruano observó que esto no era así: se podían reproducir e, incluso, hibridar con sus contrapartes no transgénicas.

Fuente: Glofish.
Cosas como esta hacen que uno sea más riguroso al momento de evaluar la liberación de un transgénico en el ambiente, ya sea para reducir las poblaciones de vectores de enfermedades o restaurar un ecosistema contaminado (biorremediación). En el ambiente, casi ningún factor puede ser controlado, por lo que la estrategia de biocontención empleada debe funcionar eficientemente. Lo que menos queremos es que el transgénico se disemine sin control; sino que cumpla la función para lo que fue desarrollado.
Aminoácidos sintéticos
Toda la vida en la Tierra habla el mismo lenguaje. No es la música ni el amor. Es el código genético.
El ‘manual de instrucciones’ de un ser vivo (conocido también como genoma o material genético) está formado por cuatro componentes: A, T, C y G (nucleótidos), los cuales están dispuestos en secuencias específicas de acuerdo al gen. Los genes producen cada de las moléculas funcionales de las bacterias, plantas, animales, hongos, etc., del planeta llamadas proteínas. Estas, a su vez, son cadenas formadas por 20 componentes diferentes llamados aminoácidos.
La pregunta es: ¿cómo podemos codificar una cadena de aminoácidos en base a la información almacenada en cuatro letras? Si cada nucleótido codificara un aminoácido, solo nos alcanzaría para cuatro. Si los combinamos en pares (AT, AG, CC, etc.) nos alcanzaría para 16. Pero si los tomamos en tripletes (ATT, GCA, TCG, etc.), nos alcanzaría para 64. Es así que el código genético se basa en tripletes para codificar los 20 aminoácidos de acuerdo a la siguiente tabla:
NOTA: Las T (timina) se cambian por U (uracilo) durante el paso de ADN a ARN mensajero (transcripción) previo a la lectura del código genético.
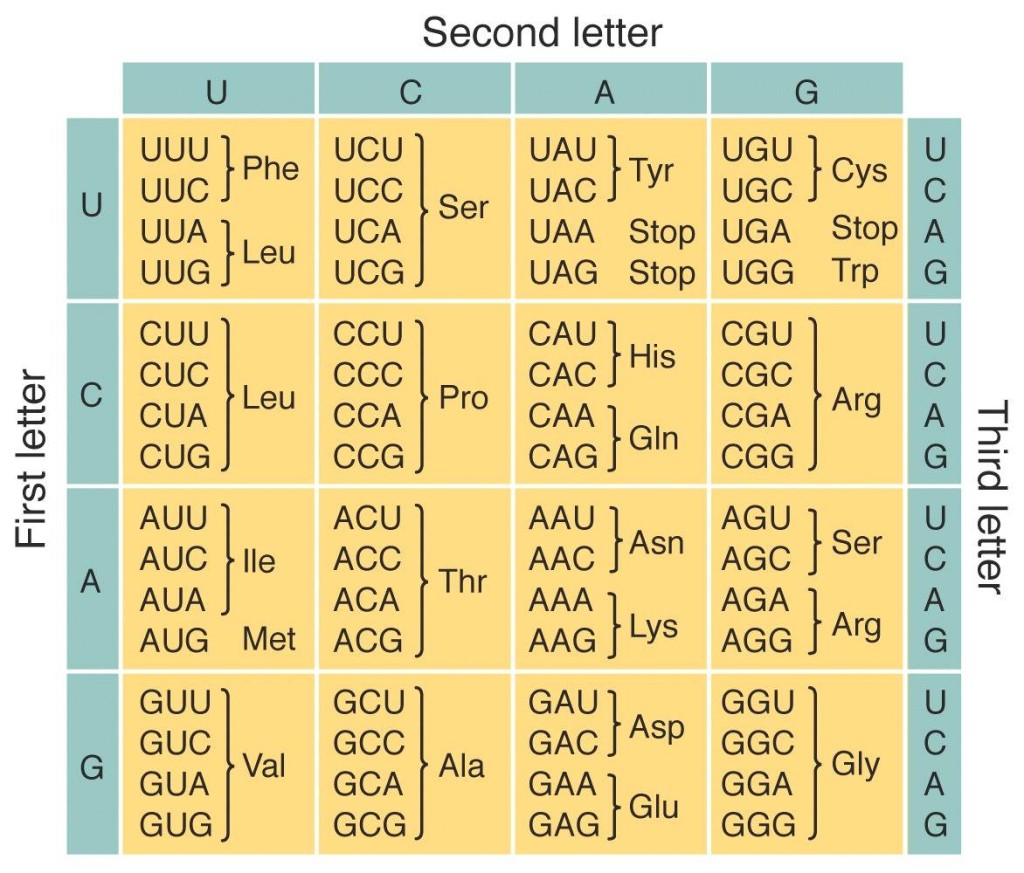
El código genético. A cada triplete se le llama codón. Existen aminoácidos que pueden ser codificados por más de un codón.
Pueden ver que hay tres tripletes que no codifican un aminoácido sino una parada o ‘STOP‘. Estos tripletes (UAA, UAG y UGA) indican dónde termina la secuencia genética para que no sigan incorporando más aminoácidos por las puras.
Lo que hicieron los investigadores de Yale, liderados por el Dr. Farren Isaacs, fue usar una cepa de Escherichia coli que no tiene el triplete TAG —el codón UAG— dentro de su genoma, para modificarlo y que codifique un aminoácido sintético no disponible en la naturaleza llamado p-acetil-L-fenilalanina (simplemente, pAcF). Es decir, recodificaron el codón UAG de ‘STOP’ por el del pAcF.
Entre uno y cuatro tripletes TAG fueron introducidos en medio de 22 genes esenciales de E. coli. De esta manera, si pAcF no estaba presente en el medio de cultivo, las bacterias no podrían expresar estos genes esenciales y morían.

Se incorpora el triplete TAG —que codifica el aminoácido sintético (sAA)— dentro de los genes esenciales. Si este sAA no está presente en el medio de cultivo, la proteína esencia sale incompleta, no es funcional y la bacteria muere. Fuente: Isaacs et al. Nature (2015).
Isaacs y su equipo además demostraron que ninguna de las 100.000 millones de bacterias genéticamente recodificadas no pudo vivir más de siete días sin el aminoácido sintético en el medio de cultivo. En otras palabras, la biocontención era extraordinariamente eficiente.
En un estudio paralelo, el equipo de Harvard liderados por el Dr. George Church (quien fuera asesor posdoctoral de Farren Isaacs antes de que migrara a Yale), desarrollaron una estrategia diferente para alcanzar el mismo objetivo. Utilizaron la misma cepa de E. coli que empleó el equipo de Isaacs pero recodificaron el codón UAG para que incorpore otro aminoácido sintético: la L-4,4′-bifenilalanina (simplemente, bipA).
Por sus características químicas, este aminoácido sintético puede incorporarse en el núcleo de las proteínas y mantenerlas funcionales. Usando poderosas herramientas computacionales, diseñaron y evaluaron la función de 112 proteínas esenciales que incorporaban el bipA en su estructura central. Al igual que en el caso anterior, sin este aminoácido, la bacteria genéticamente recodificada muere.
Y no solo eso, estas bacterias tampoco pueden adquirir mutaciones o recibir genes de otras bacterias para poder sobrevivir en ausencia de los aminoácidos sintéticos, pues el candado no está en los genes sino en el ‘lenguaje de programación’. Es como tratar de instalar una aplicación de Android (código genético estándar) en iPhone (código genético modificado).
Futuras aplicaciones
Estos OGR tienen un gran potencial en la biorremediación. Podemos desarrollar bacterias con genes que permitan degradar el petróleo de los océanos, los residuos químicos de las fábricas o capturar los metales pesados de los relaves mineros. Y, una vez que cumplan con su función, les damos las gracias y dejamos de proveerles del aminoácido sintético para eliminarlos del ecosistema.

Fuente: Wikimedia Commons.
Por otro lado, estos OGR serían inmunes al ataque de muchos virus pues poseen un código genético diferencial. Mientras que en los virus el codón TGA indica el fin de la secuencia de una de sus proteínas esenciales, la bacteria —quien es el que pone la maquinaria para expresar los genes del virus— no lo ve así y le incorpora el aminoácido sintético, creando una proteína viral defectuosa. Esto sería importante para la producción de fármacos o compuestos alimenticios que, en algunas ocasiones, se echan a perder grandes lotes de producción a causa de la infección por un virus.
Finalmente, una aplicación un poco más compleja es en el desarrollo de plantas transgénicas. Estas no podrían diseminarse a menos que se les provea del aminoácido sintético. Sin embargo, antes se debe evaluar la inocuidad de este aminoácido y de las proteínas que constituye.
Por ahora, el futuro más prometedor está en los microorganismos. Church e Isaacs siguen trabajando para recodificar otros codones y no solo generar medidas de biocontención más eficientes; sino proteínas que incorporen nuevos aminoácidos con funciones completamente nuevas. Este es un paso más dentro de la biología sintética.

:quality(75)/2.blogs.elcomercio.pe/service/img/expresiongenetica/autor.jpg)