Biología sintética: Ampliando el alfabeto genético
El miércoles pasado, Nature publicó un revolucionario estudio que ha sido cubierto por diferentes medios de comunicación científica. Las mejores notas las pueden encontrar aquí, aquí, aquí y aquí (en inglés) y aquí y aquí (en español). Aprovechando toda esta rica información disponible, expondremos el tema de una forma más digerible.

Expansión del alfabeto del ADN: A, T, C, G, X e Y. Fuente: Synthorx.
Un poco de biología molecular básica
En un anterior post hablamos algo sobre el ADN, una molécula maravillosa que contiene toda la información requerida para “construir” un ser vivo. Es un manual de instrucciones que le indica a la célula cómo debe crecer, multiplicarse, diferenciarse y cumplir con una función específica, ya sea de forma individual —en el caso que sea una bacteria o una ameba— o formando parte de un determinado tejido en un organismo mucho más complejo.
El ADN está compuesto de cuatro moléculas llamadas nucleótidos, conformados por una base nitrogenada que puede ser adenina (A), guanina (G), timina (T) y citocina (C), unidas a un azúcar llamado ribosa y a un átomo de fósforo con varios oxígenos alrededor. En el ADN, los nucleótidos se disponen en pares: A con T y G con C, uno sobre otro, formando una larga cadena helicoidal similar a una escalera de caracol.
Lo hermoso de esta molécula es que nos permite transmitir la información genética a los descendientes de manera fidedigna. Basta con abrir la cadena de ADN por la mitad —como si fuera una cremallera— para poder replicarla (Figura 1).
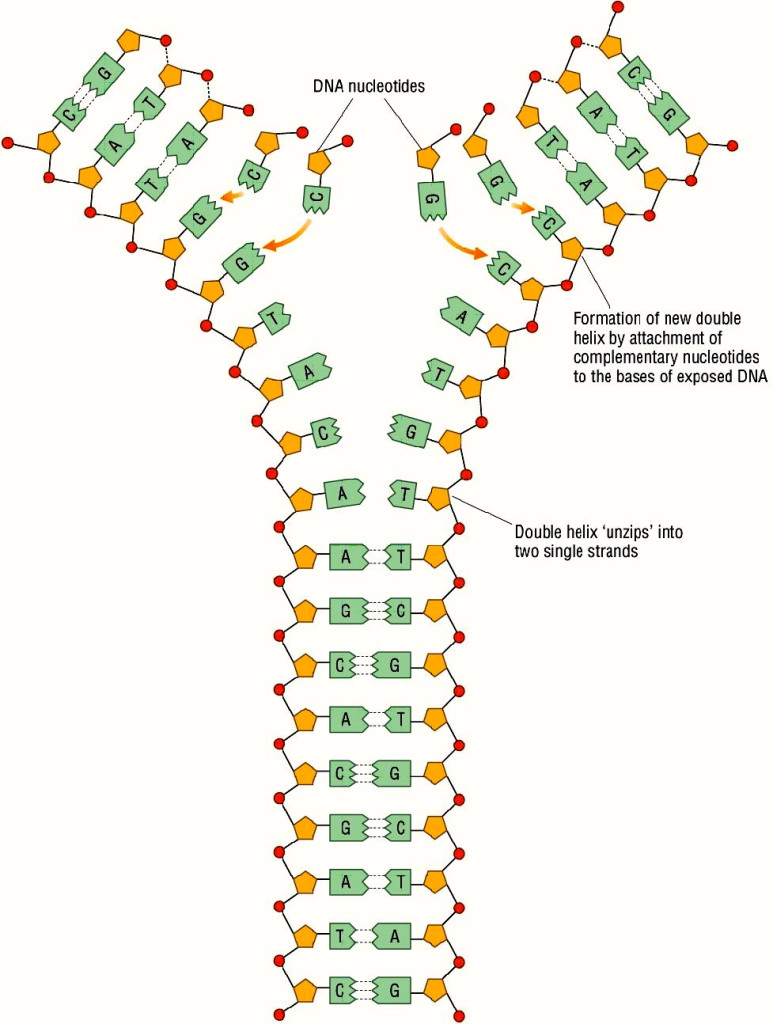
Fig 1. Replicación de ADN en función a la complementariedad de las bases. Fuente.
¿Y cómo hace el ADN para contener y transmitir información? Pues los cuatro nucleótidos forman un código de cuatro letras (A, T, C y G), por ejemplo, …ATTAGGCGATA… o …GGTACGGAAATTAC… Las posibilidades son infinitas.
Una determinada secuencia de nucleótidos codificará un gen. En el ADN de un organismo pueden haber miles de genes, así como también, secuencias que no codifican nada o que solo sirven para regular la expresión de los genes (interruptores genéticos). Finalmente, el producto de expresión de los genes son las proteínas.
Las proteínas son largas cadenas de aminoácidos que se ordenan en función a la secuencia de los genes. Los seres vivos necesitan al menos 20 aminoácidos diferentes para formar todas las proteínas necesarias para cumplir con sus funciones. Entonces, la pregunta es ¿cómo codificamos los 20 aminoácidos usando un código basado en cuatro letras?
Si el código utilizara una letra a la vez, sólo podría codificar cuatro aminoácidos. Si utilizara una combinación de dos letras (AT, CG, AA, CC, GA…) se podría codificar 16 aminoácidos —aún nos quedaríamos cortos. Si utilizara una combinación de tres letras (ATT, ACG, CGG, CTG…) se podría codificar hasta 64 aminoácidos. Es así que la combinación de tres letras codifican los 20 aminoácidos necesarios para la vida (Figura 2).
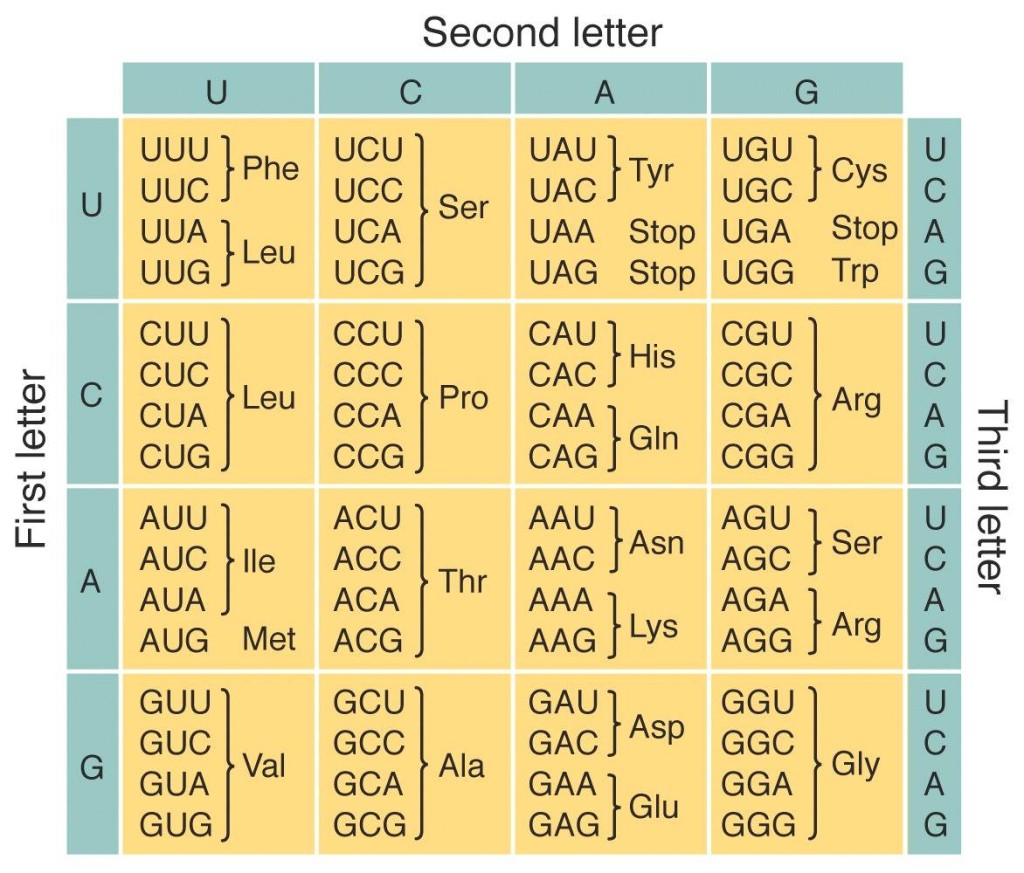
Fig 2. El código genético. Fuente.
Como pueden ver en el código genético, un aminoácido puede ser codificado por más de un triplete. Además, en el código genético vemos una U en vez de una T. Esto se debe a que los genes en el ADN no pueden ser traducidos directamente a proteínas, requieren de un intérprete llamado ARN mensajero, muy similar al ADN, pero que sólo tiene una hebra y donde la timina (T) es reemplazada por el uracilo (U).
Dos letras más…
Que el ADN esté formado por A, T, C y G (U en el caso del ARN) puede ser producto de un accidente químico y evolutivo más que de una necesidad funcional porque se ha demostrado que otros nucleótidos similares “no naturales” también pueden ser incorporados en el ADN.
En 1989, el Dr. Steven Benner se convirtió en el primer científico que lograba incorporar dos nuevos nucleótidos —isómeros de la guanina y citosina— en moléculas de ADN y ARN. Si bien el experimento se realizó en tubos de ensayo y no en un organismo vivo, esto marcaba el inicio en la búsqueda de la expansión del alfabeto genético.
Los esfuerzos por ampliar el alfabeto genético mediante la adición de pares de bases no naturales prometen ampliar las aplicaciones biotecnológicas disponibles para el ADN, además de ser un primer paso esencial hacia la expansión del código genético. En el año 2008, el Dr. Floyd Romesberg emparejó una lista de 60 nucleótidos no naturales candidatos con el fin de encontrar pares de base compatibles con la vida. De las 3600 combinaciones posibles, sólo una parecía prometedora. Los nuevos nucleótidos se llamaban d5SICS y dNaM, pero de cariño les dicen X e Y.
Un año después, Romesberg y su equipo lograron transcribir un ADN con los seis nucleótidos —A, T, C, G, X e Y— a ARN (Figura 3) y, en el 2012, lograron replicarlo utilizando las mismas enzimas que se encuentran en los seres vivos. Si bien ambos experimentos fueron realizados in vitro, o sea, en un tubo de ensayo, replicarlo en un organismo vivo estaba cada vez más cerca.

Fig 3. Transcripción con el alfabeto genético extendido. Fuente: Jun Seo, et al. (2009).
Y es así como llegamos al artículo publicado el miércoles en Nature. El equipo del Dr. Romesberg logró incorporar un nuevo par de bases (X:Y) en un microorganismo y este lo pudo replicar y mantener por varias generaciones. ¿Cómo lo hicieron?
El primer paso fue incorporar incorporar las bases X:Y en el ADN de un organismo fácil de criar y modificar. Para ello usaron a la Escherichia coli, la bacteria más estudiada de la ciencia. Para facilitar el trabajo no insertaron X:Y directamente en el genoma de la bacteria sino en una pequeña porción de ADN llamado plásmido, que tiene la capacidad de replicarse independientemente.
El segundo paso era hacer que las bases X e Y ingresen a la bacteria. Normalmente no lo pueden hacer a menos que exista “algo” que las transporte a través de la membrana celular. Ese algo se obtuvo de una alga. Se trataba de una proteína transportadora que forma un canal a través de la membrana para permitir el paso de X e Y.
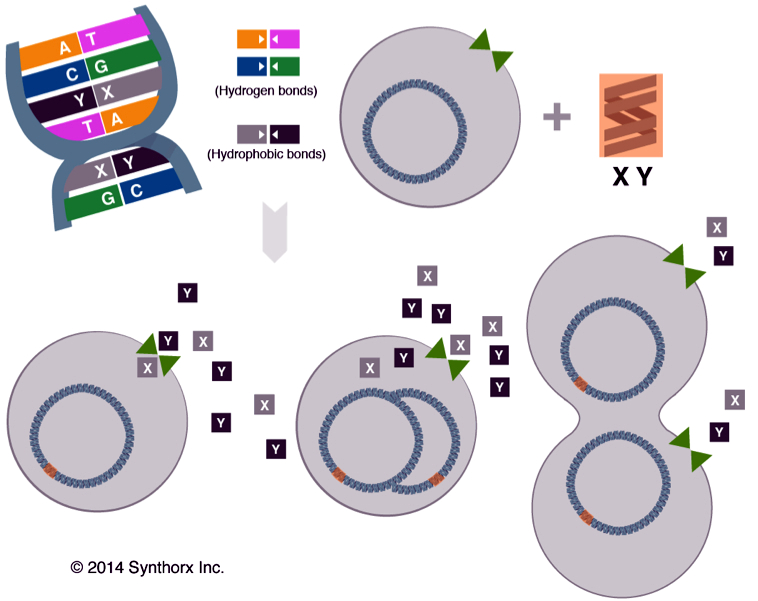
Fig 4. Procedimiento para generar bacteria con el alfabeto genético extendido. Adaptado de Synthorx.
Y así fue como lograron que la bacteria no sólo viva con un ADN con un par de bases completamente nuevo, sino que trasmita esta misma información a sus descendientes por varias generaciones. Además, si no se le proveía de X e Y en el medio de cultivo, las bacterias lo eliminaban de su ADN y volvían a su estado natural de cuatro nucleótidos. Esto podría servir como una medida de seguridad para evitar que se diseminen por el mundo.
Entonces, con un alfabeto de seis letras tenemos la posibilidad de expandir el código genético para poder incorporar otros aminoácidos distintos a los 20 naturales (Figura 5). Podríamos generar nuevas proteínas con nuevas funciones, sin precedentes en la naturaleza, para aplicarlos en la medicina, en la industria, en la descontaminación del ambiente, etc. Las aplicaciones son infinitas.
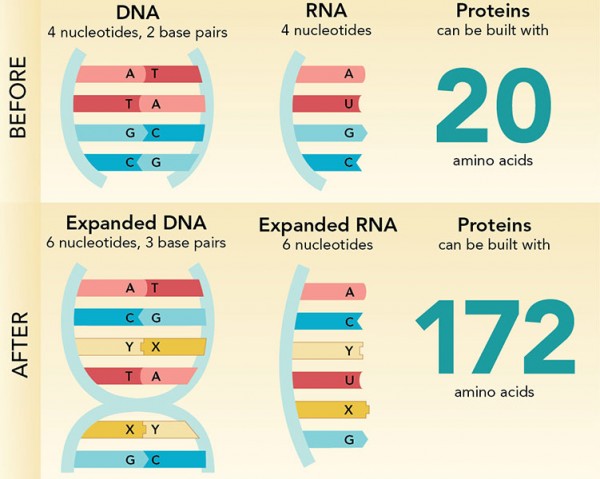
Fig 5. Expansión del código genético. Fuente: Science NOW.
La importancia de este estudio radica en que es la primera vez en que un nuevo par de bases puede ser incorporado y mantenido en un organismo vivo. Es un primer paso, muy importante, pero aún queda mucho camino por recorrer hasta aprovechar todas las posibilidades que puede ofrecer la biología sintética.
El siguiente paso debe ser lograr que el ADN con seis nucleótidos se logre transcribir a ARN mensajero, luego que pueda ser traducido a proteína. Una vez alcanzado estos objetivos se podría pensar en diseñar nuevos ARN de transferencia con nuevos aminoácidos y ver si pueden formar proteínas. Y después de lograr todo esto, diseñar proteínas con funciones novedosas. Esperemos que en los próximos años todo vaya por buen camino.
Referencia:
Malyshev DA, Dhami K, Lavergne T, et al. A semi-synthetic organism with an expanded genetic alphabet. Nature. (2014) doi: 10.1038/nature13314

:quality(75)/2.blogs.elcomercio.pe/service/img/expresiongenetica/autor.jpg)