Evolución de la resistencia a antibióticos en video
Doce días… Ese fue el tiempo que le tomó a una bacteria volverse cien mil veces más resistente a uno de los antibióticos más usados hoy en día —la ciprofloxacina. Y una decena de días para volverse diez mil veces más resistente a la trimetroprima. Estos fueron los resultados de un ingenioso experimento realizado por investigadores del departamento de biología de sistemas de la Universidad de Harvard.
§§§
Uno de los errores más frecuentes que cometen las personas es automedicarse. Si sienten un dolor de estómago, van por un Bactrim® (trimetroprima más sulfametoxazol). Para un dolor de garganta, se toman una pastilla de amoxicilina con ácido clavulánico. Si les sale mucho acné, van por la doxiciclina. Todos son antibióticos y se venden bajo receta médica. Sin embargo, hay farmacias y boticas que las venden libremente o uno las tiene en su casa porque le sobraron de alguna infección anterior.
El problema radica en que tomamos antibióticos cuando no los necesitamos (por ejemplo, en un cuadro gripal), no tomamos las dosis adecuadas o no terminamos con el tratamiento que el médico nos prescribe porque creemos sentirnos bien a los pocos días (de ahí que nos sobran para futuras ocasiones). Esto provoca que las bacterias infecciosas se vuelvan cada vez más resistentes a ellos y que cada año mueran unas 700 mil personas a causa de este problema.
El modo de vida de las bacterias se resume de la siguiente manera: cuando hay suficientes nutrientes en el medio, se multiplican vertiginosamente hasta que los recursos son escasos. Cuando llegan a este punto, entran en un estado de latencia o buscan la forma de migrar y adaptarse a un nuevo entorno en vez de competir con sus vecinas por lo poco que queda. Este comportamiento fue aprovechado por un grupo de investigadores de la Universidad de Harvard, liderados por el Dr. Roy Kishony, para ver en tiempo real cómo evoluciona la resistencia a dos antibióticos en la bacteria E. coli.
Lo que hicieron los investigadores fue poner un medio de cultivo rico en nutrientes —y teñido de negro— en una bandeja acrílica de 120 por 60 centímetros (algo así como una placa Petri gigante), dividido en nueve regiones. Cada región tenía diferentes concentraciones de antibiótico, empezando con cero en los extremos y aumentando a razón de diez hacia el centro.
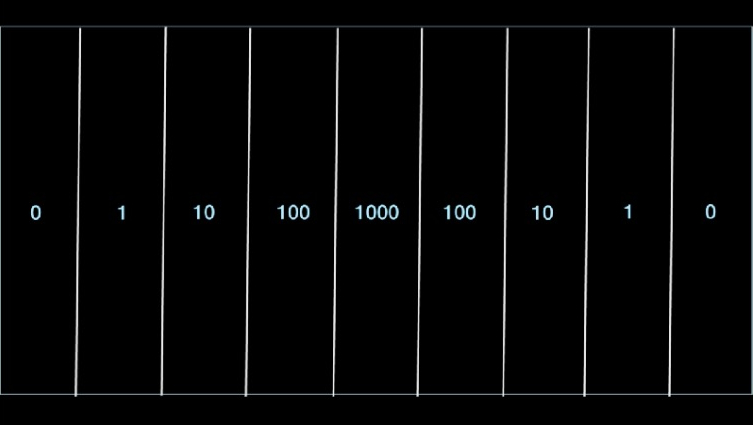
Placa diseñada para el presente experimento. Los números representan las proporciones de antibiótico utilizado en cada región.
Para el caso de la ciprofloxacina se usó concentraciones de 0, 20, 200, 2000 y 20.000 unidades MIC (concentración mínima de antibiótico requerido para inhibir el crecimiento de las bacterias); mientras que para la trimetroprima, 0, 3, 30, 300 y 3000 unidades MIC. Las bacterias fueron inoculadas a cada extremo de la placa y una cámara ubicada en la parte superior tomó fotos cada cierto tiempo con el que se hizo un time-lapse.
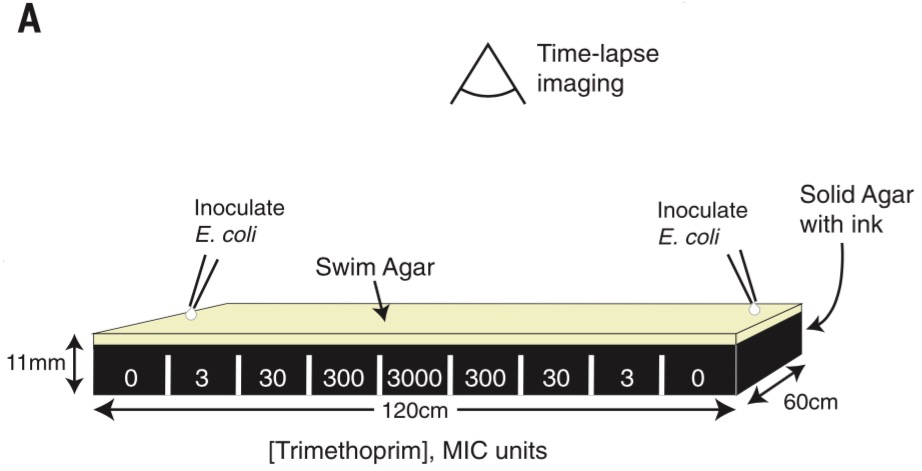
Esquema de la placa usada en el experimento. Fuente: Baym et al. (2016).
El resultado fue espectacular, solo vean el siguiente video:
The Evolution of Bacteria on a “Mega-Plate” Petri Dish from Harvard Medical School on Vimeo.
Como pueden ver, la colonia bacteriana (mancha blanca) empieza a extenderse desde el punto de inoculación hasta abarcar el primer sector del medio de cultivo que está libre de antibiótico. Luego se detiene. Es como si hubiera una pared delante de ella. La primera dosis de antibiótico inhibe su crecimiento, aunque no por mucho tiempo. El primer mutante resistente aparece y empieza a diseminarse hasta alcanzar el siguiente sector con una concentración 10 veces mayor de antibiótico.

Aparición de las primeras bacterias resistentes al antibiótico. Fuente: Baym et al. (2016).
Este proceso se repite una y otra vez hasta que la colonia bacteriana alcanza el sector central con una concentración diez mil veces superior a la inicial. En el caso de la trimetroprima tardó solo diez días, mientras que para la ciprofloxacina lo hizo en doce días.
Los investigadores tomaron muestras en diferentes puntos del medio de cultivo con trimetroprima para realizarles un análisis genético. Los resultados mostraron la aparición de diversas mutaciones en el gen que codifica la enzima responsable de la replicación del ADN de la bacteria (dnaQ). Esta enzima es inhibida por la acción de antibiótico. Otro dato importante es que las mutaciones en el gen dnaQ aparecieron al menos seis veces independientemente en distintos puntos del medio de cultivo.
Utilizando los datos genéticos, los investigadores también pudieron rastrear los linajes mutantes más resistentes desde su origen, tal como muestra la siguiente imagen:

Las líneas y puntos rastrean el origen de los linajes mutantes más resistentes. Fuente: Baym et al. (2016).
Finalmente, en un último experimento demostraron que las bacterias tardan mucho más en adaptarse a la mayor concentración de antibiótico si no hay concentraciones intermedias más tolerables (es decir, si pasa directamente de 0 a 3000 unidades MIC). Asimismo, las bacterias que se encontraban en la zona intermedia de cada sector se adaptaban más rápido a una mayor concentración de antibiótico que las bacterias ubicadas en el límite de cada sector.

Cuando hay una concentración intermedia menor a la máxima (3000 MIC), la adaptación tarda menos horas. Fuente: Baym et al. (2016).
Sin dudas es un experimento muy ilustrativo aunque los investigadores aclaran que esto no simula las condiciones naturales o clínicas que se presentan. Sin embargo, esta metodología muestra aspectos claves de la evolución de la resistencia a los antibióticos que puede ser aplicado a diferentes organismos, por ejemplo, para identificar las mutaciones que aparecen y ver la forma de contrarrestarlas.
Referencia:
![]() Baym, M., Lieberman, T., Kelsic, E., Chait, R., Gross, R., Yelin, I., & Kishony, R. (2016). Spatiotemporal microbial evolution on antibiotic landscapes Science, 353 (6304), 1147-1151 DOI: 10.1126/science.aag0822
Baym, M., Lieberman, T., Kelsic, E., Chait, R., Gross, R., Yelin, I., & Kishony, R. (2016). Spatiotemporal microbial evolution on antibiotic landscapes Science, 353 (6304), 1147-1151 DOI: 10.1126/science.aag0822

:quality(75)/2.blogs.elcomercio.pe/service/img/expresiongenetica/autor.jpg)